MinION で得られた DNA 配列の確認
今回、ゲノム弁当から MinION で読んだ DNA 配列データは、デスクトップの MinION フォルダ以下に置いてあります。このうち、1_hakusai と 5_hiyokomame がゲノムをまるごとシーケンスしたデータ、2_ninjin, 3_tsukemono, 4_takikomigohan, 6_tomato が PCR をかけて rbcL 遺伝子領域だけを増幅したものになっています。
各フォルダを開いていくと、それぞれに fast5_pass, fast5_fail, fastq_pass, fastq_fail および sequencing_summary フォルダが含まれていることが分かります。この中で、今回の解析に使うのは fastq_pass だけです。なお fast5 とは HDF5 という複雑かつ大規模なデータを格納できるファイル形式を用いた、MinION シーケンサーの出力する生データになります。このままでは取り扱いが難しいため、DNA配列と配列のクオリティを4行ずつに束ねた FASTQ 形式に変換されたものを利用します(以前は poretools を使って自分で変換する必要がありましたが、今の MinION についてくる MinKNOW ソフトウェアは自動で FASTQ ファイルを生成してくれるようになっています)。また、fast5_fail と fastq_fail は何らかの理由で解読に失敗したデータなので割愛します。また前述のように fast5_pass は fastq_pass の元になった HDF5 形式のバイナリデータのため今回は使いません。最後の sequencing_summary フォルダにはシーケンシングの実行ログが記録されています。
MinION
|-- 1_hakusai
| `-- 20190301_1336_MN21603_FAK44689_d305e2cb
| |-- fast5_fail
| |-- fast5_pass
| |-- fastq_fail
| |-- fastq_pass
| | |-- FAK44689_c23e7ecf3758750d90215871e80bac6c68c94336_0.fastq
| | |-- FAK44689_c23e7ecf3758750d90215871e80bac6c68c94336_1.fastq
| | |-- FAK44689_c23e7ecf3758750d90215871e80bac6c68c94336_2.fastq
| | |-- FAK44689_c23e7ecf3758750d90215871e80bac6c68c94336_3.fastq
| | |-- FAK44689_c23e7ecf3758750d90215871e80bac6c68c94336_4.fastq
| | |-- FAK44689_c23e7ecf3758750d90215871e80bac6c68c94336_5.fastq
| | `-- FAK44689_c23e7ecf3758750d90215871e80bac6c68c94336_6.fastq
| `-- sequencing_summary
|-- 2_ninjin
| `-- 20190301_1337_MN21648_FAK39813_5aa5b150
| |-- fast5_fail
| |-- fast5_pass
| |-- fastq_fail
| |-- fastq_pass
| | |-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_0.fastq
| | |-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_1.fastq
| | |-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_2.fastq
| | |-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_3.fastq
| | |-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_4.fastq
| | |-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_5.fastq
| | |-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_6.fastq
| | |-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_7.fastq
| | `-- FAK39813_540392b51a4e938730e82c883f056985d9abc2c6_8.fastq
| `-- sequencing_summary
|-- 3_tsukemono
| `-- 20190301_1332_MN20345_FAK42049_c533ace0
| |-- fast5_fail
| |-- fast5_pass
| |-- fastq_fail
| |-- fastq_pass
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_0.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_1.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_2.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_3.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_4.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_5.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_6.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_7.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_8.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_9.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_10.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_11.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_12.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_13.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_14.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_15.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_16.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_17.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_18.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_19.fastq
| | |-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_20.fastq
| | `-- FAK42049_d71bfefd1e345fe72563b61dc5ba7bd077d6c347_21.fastq
| `-- sequencing_summary
|-- 4_takikomigohan
| `-- 20190301_1333_MN23533_FAK41937_db91d304
| |-- fast5_fail
| |-- fast5_pass
| |-- fastq_fail
| |-- fastq_pass
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_0.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_1.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_2.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_3.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_4.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_5.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_6.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_7.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_8.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_9.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_10.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_11.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_12.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_13.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_14.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_15.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_16.fastq
| | |-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_17.fastq
| | `-- FAK41937_0dc513f647a247ce16e31f65e078051671d5dfb1_18.fastq
| `-- sequencing_summary
|-- 5_hiyokomame
| `-- 20190301_1334_MN21672_FAK41983_f5af709e
| |-- fast5_fail
| |-- fast5_pass
| |-- fastq_fail
| |-- fastq_pass
| | |-- FAK41983_305c4f329d6b4d936004adebf09704b49e0eb992_0.fastq
| | |-- FAK41983_305c4f329d6b4d936004adebf09704b49e0eb992_1.fastq
| | |-- FAK41983_305c4f329d6b4d936004adebf09704b49e0eb992_2.fastq
| | |-- FAK41983_305c4f329d6b4d936004adebf09704b49e0eb992_3.fastq
| | `-- FAK41983_305c4f329d6b4d936004adebf09704b49e0eb992_4.fastq
| `-- sequencing_summary
`-- 6_tomato
`-- 20190301_1337_MN18551_FAK44751_422dd586
|-- fast5_fail
|-- fast5_pass
|-- fastq_fail
|-- fastq_pass
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_0.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_1.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_10.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_2.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_3.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_4.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_5.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_6.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_7.fastq
| |-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_8.fastq
| `-- FAK44751_adba464afe20b0a6c54cb00da954f68823f4c473_9.fastq
`-- sequencing_summary
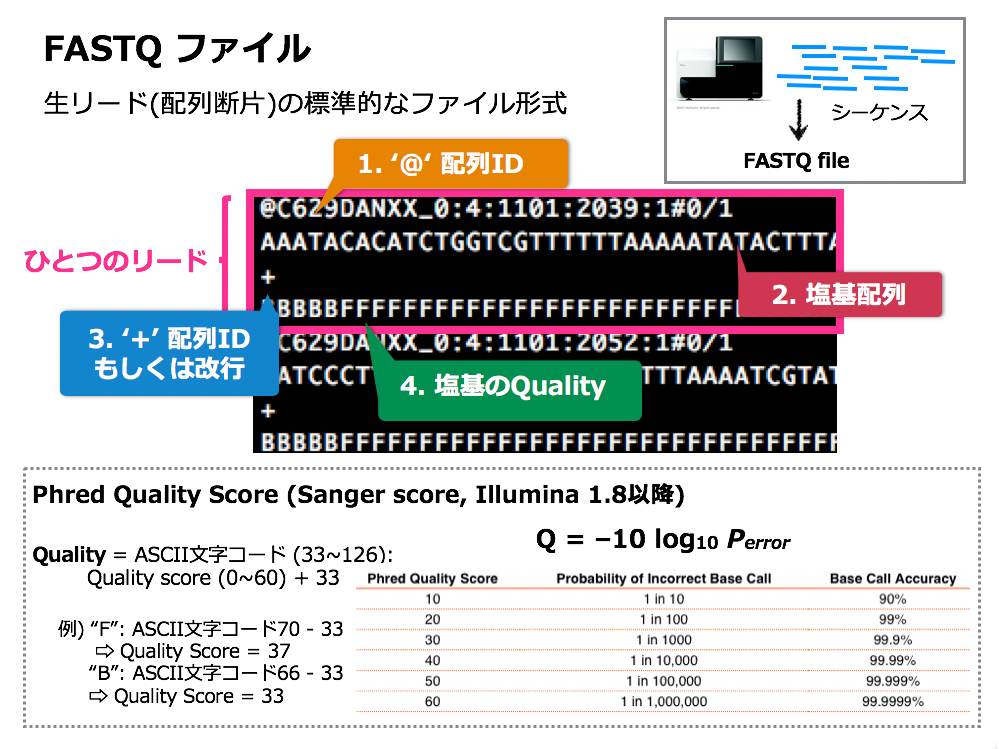
FASTQファイルの構造
FASTQ ファイルは、1つの DNA 配列につき4行ごとに1セットとなっています。以下の図にあるように、塩基配列は2行目に書かれています。
FASTQ ファイルを1つにまとめる
MinION の出力する FASTQ ファイルは最大で 4000 配列ごとに1ファイルになるようです。複数ファイルに分かれたままでは扱えないツールもあるため、今回はそれぞれの実験ごとに1つずつの FASTQ ファイルにまとめてから利用することにします。cat コマンドで複数のファイルを1つに繋げることができます。ここでは、つなげたファイルをデスクトップの GenomeBento/samples/ フォルダに置いておくことにします。
cat ~/Desktop/MinION/1_hakusai/2019*/fastq_pass/*.fastq > ~/Desktop/GenomeBento/samples/1_hakusai.fastq
cat ~/Desktop/MinION/2_ninjin/2019*/fastq_pass/*.fastq > ~/Desktop/GenomeBento/samples/2_ninjin.fastq
cat ~/Desktop/MinION/3_tsukemono/2019*/fastq_pass/*.fastq > ~/Desktop/GenomeBento/samples/3_tsukemono.fastq
cat ~/Desktop/MinION/4_takikomigohan/2019*/fastq_pass/*.fastq > ~/Desktop/GenomeBento/samples/4_takikomigohan.fastq
cat ~/Desktop/MinION/5_hiyokomame/2019*/fastq_pass/*.fastq > ~/Desktop/GenomeBento/samples/5_hiyokomame.fastq
cat ~/Desktop/MinION/6_tomato/2019*/fastq_pass/*.fastq > ~/Desktop/GenomeBento/samples/6_tomato.fastq
なお、ディレクトリ名やファイル名に * を含めると、そこにある全てのファイルにマッチするので、長い名前を省略したり、同じ名前で始まるファイルをまとめて指定することができます。また途中まで入力して Tab キー(Mac だと ->| キー)を押すと、長いファイル名も自動的に補完されるので、タイプ量を減らすことができます。
BLAST による類似配列検索
TODO: 追記する